Virale Diagnostik
Virale Diagnostik
Cube Dx bietet eine schnelle aber dennoch umfangreiche Analyse von SARS-Cov-2-Mutationen positiver Proben. Dies ermöglicht einerseits eine rasche Stratifizierung der Patienten und andererseits eine schnelle Reaktion auf neue Varianten, die beispielsweise in einer Region zum ersten Mal auftreten. Die detaillierte Mutationsübersicht ermöglicht eine gezielte Auswahl von besonders relevanten Proben für die weitere Sequenzierung. Speziell die Mutationen in der Receptor-Binding-Domain (RBD) des S-Gens sind klinisch relevant, da diese Mutationen die Effektivität von Impfstoffen und den Krankheitsverlauf beeinflussen können.
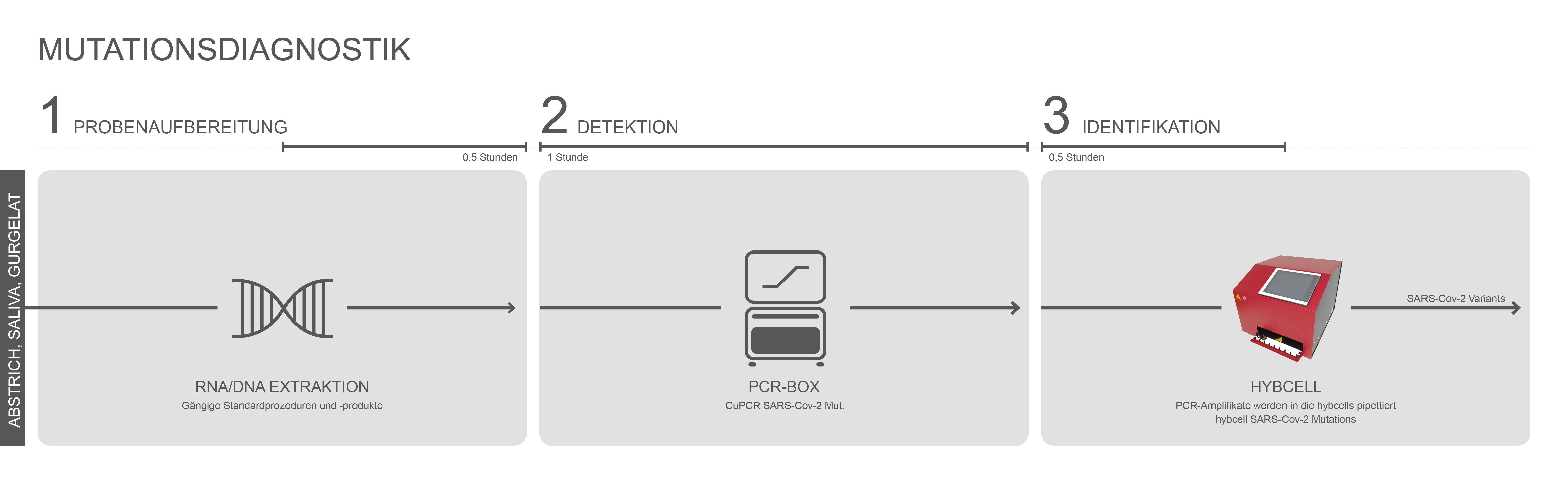
compact sequencing ist ein zweistufiger Prozess. Der erste Schritt ist dabei eine einfache RT-PCR-Reaktion (die einen Abschnitt des S-Gens amplifiziert). Dieser Schritt liefert einzelsträngige, fluoreszenzmarkierte DNA für den zweiten Schritt und ermöglicht eine quantitative Schätzung der viralen Belastung. Der zweite Schritt ist die Identifizierung von Mutationen und die Datenanalyse im hyborg. Die Kombination aus quantitativer Abschätzung und Nachweis auch mehrerer Varianten (Mischungen) parallel ist einzigartig. Die einfache Handhabung, das breite Mutationspanel und die automatisierte Analyse von Varianten basierend auf compact sequencing reduzieren den Personalbedarf für die Mutationsdiagnostik drastisch. Der gesamte Prozess dauert weniger als zwei Stunden.
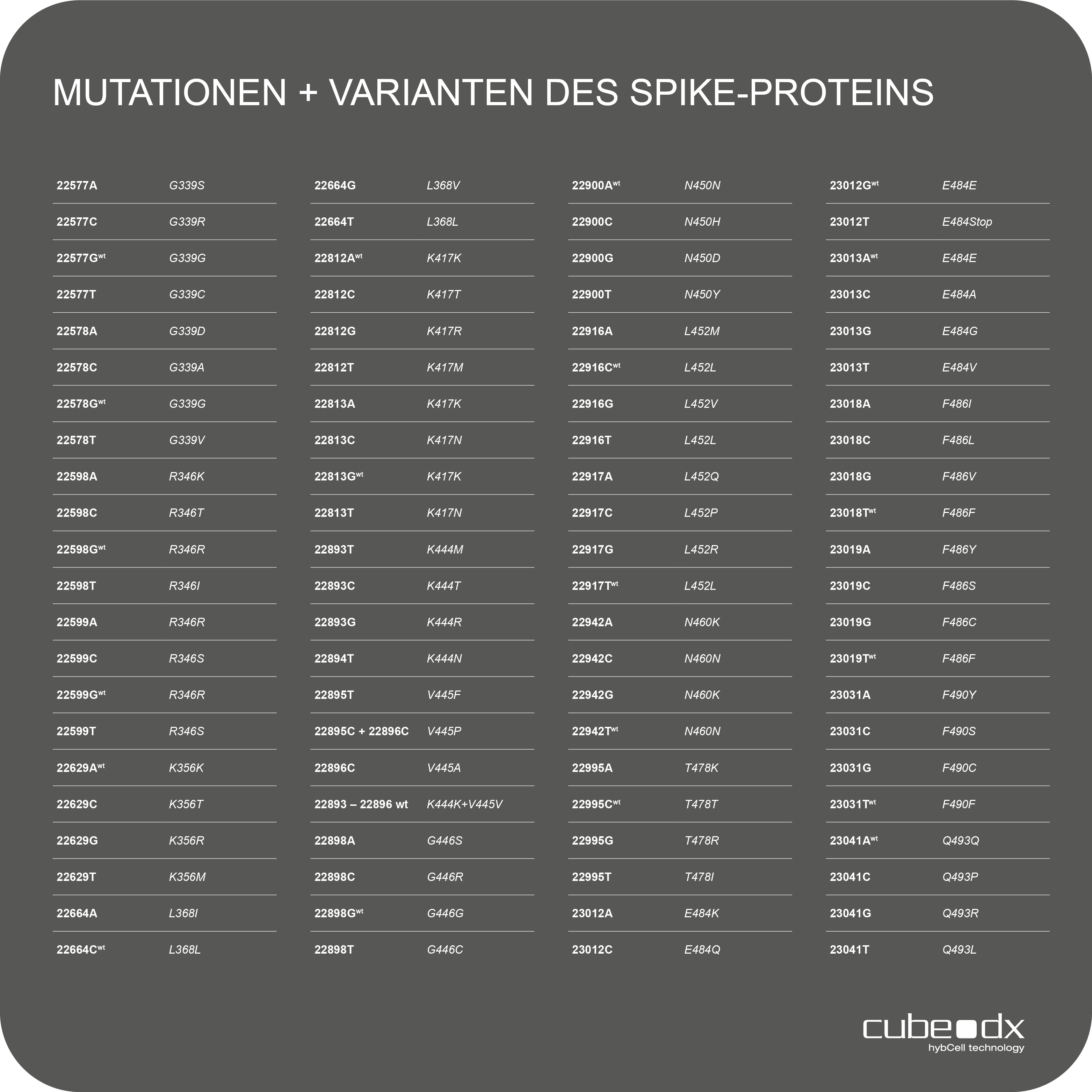
88 Mutationen / Alle relevanten Omicron-Varianten
EPIDEMIOLOGISCHE ÜBERWACHUNG VON SARS-COV-2-VARIANTEN:
Cube Dx‘ schnelle und umfassende Mutationsanalyse von SARS-Cov-2 identifiziert sehr rasch Phenotypen mit geänderten Mutationsmustern und ermöglicht so ein rasches Handeln, um neue Varianten einzudämmen.
Die RT-PCR Reaktion (CuPCR SARS-Cov-2 Mut.) ist der erste Schritt der Mutationsanalyse und basiert auf einer „One-Step“ Reaktion von reverser Transkription (RT) kombiniert mit der PCR zur Amplifikation der RNA von SARS-Cov-2 (S-Gen). Der Test basiert auf den Prinzipien der TaqMan-PCR und verwendet einen Fluoreszenzfarbstoff (JOE) zum Nachweis der Amplifikation.
Positive Amplikons werden sofort auf relevante Mutationen in der Rezeptorbindungsdomäne (RBD) des S-Gens von SARS-Cov-2 mit hybcell SARS-Cov-2 Mutations unter Verwendung des proprietären compact sequencing von Cube Dx untersucht. In nur 15 Minuten können 88 SNPs (Single Nucleotide Polymorphism = Mutationen) durch die Identifizierung von Basen (A, C, G, T) der +ssRNA an spezifischen Nukleotidpositionen unterschieden werden. Die sich daraus ergebenden Mutationsmuster werden von der Software mit bekannten Varianten korreliert und als solche ausgegeben.
Der Test wird regelmäßig aktualisiert, um die aktuell wichtigsten Mutationen und Varianten zu ermitteln. Es können verschiedene Proben getestet werden, z. B. Humanproben oder Abwasserproben. Der Test dient der epidemiologischen Überwachung und darf nicht als In-vitro-Diagnostikum dienen.
Eigenschaften / Vorteile:
-
Screening des S-Gens (Receptor Binding Domain)
-
Minimale "hands-on" durch unmittelbare Verwendung der positiven Amplicons, schnelles Resultat und einfache Analyse
-
Limit of detection > 20 RNA Kopien per Reaktion
-
Analyse von 88 SARS-Cov-2 Mutationen in 15 bis 20 Minuten
-
Automatisierte Auswertung der Mutationen und bekannter Varianten durch die Software
-
Paralleler Nachweis mehrerer Varianten in einer Probe (z.B. Abwasser)
-
Mit humanen Proben und Wasserproben validiert